三行代码get随机森林怎么用(随机森林代码实现)
导语:三行代码get随机森林
假期结束要开始充电学习了,今天给大家分享随机森林算法。随机森林是当下非常受欢迎的机器学习算法之一,鲁棒性好且易于使用。在数据挖掘中,我们常用它来构建分类模型,今天就来看看它在科研中的实际应用。
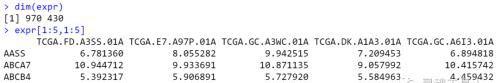
比如我们拿到了一些基因在某个疾病中的的表达谱,也有对应的样本分类,如control vs case,我们可以利用R包randomForest构建随机森林分类器,通过特征基因预测疾病分类。现在我准备了一个基因在行、样本在列的表达谱:
还有对应的表型分类:

为了适用于随机森林函数,我们先将表达谱和表型样本取交集,并将表达谱转置成基因在列样本在行的表达谱:
sam <- intersect(rownames(pheno), colnames(expr))expr <- expr[, sam]expr <- as.data.frame(t(expr))pheno <- pheno[sam, , drop=FALSE]接着设置随机种子,并构建随机森林分类模型:
set.seed(111)rf <- randomForest(x = expr, y = as.factor(pheno$tissue), importance = TRUE, proximity = TRUE, ntree = 1500, mtry = 10)我们会获得每个基因的权重(importance),用以表征基因对疾病分类的重要程度(如果分类特征是生存状态,那么就可以表征基因对预后的重要性)。利用varImpPlot()函数可以对importance可视化:
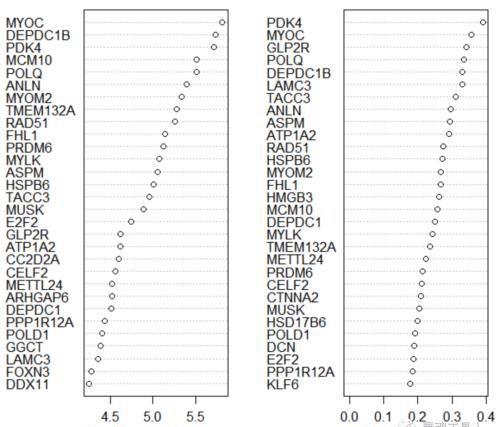
如图,随机森林给出两个指标用以评估importance,分别是MeanDecreaseAccuracy和MeanDecreaseGini。通过以上分析,会发现randomForest可以方便的构建分类器,但却不具备特征筛选功能。也就是说,它并不能像LASSO、单因素cox、多因素cox一样直接筛选特征。不过,我们可以依据特征的importance设置临界阈值筛选特征基因;也可以通过RFE等特征筛选算法进一步实现降维;另外还有一些R包已将随机森林和特征筛选打包在一起,实现一键降维,如R包Boruta、VSURF等。
今天的分享就到这了,觉得不错记得收藏转发哦!
免责声明:本站部份内容由优秀作者和原创用户编辑投稿,本站仅提供存储服务,不拥有所有权,不承担法律责任。若涉嫌侵权/违法的,请反馈,一经查实立刻删除内容。本文内容由快快网络小涵创作整理编辑!